近日,珠江水产研究所叶星研究员团队完成了大口黑鲈染色体水平基因组精细图谱的绘制和遗传解析,研究论文“Chromosome-level genome assembly for the largemouth bassMicropterus salmoidesprovides insights into adaptation to fresh and brackish water”正式发表在国际重要学术期刊《Molecular Ecology Resource》上。
大口黑鲈(Micropterus salmoides)自然分布于北美淡水流域,但在海岸咸淡水环境中也有分布,是北美水生态系统中具有重要生态学与经济学意义的黑鲈属重要成员之一。自1983年引进以来大口黑鲈已成为我国重要的养殖鱼类,2019年我国养殖产量达47.8万吨。为更好地了解大口黑鲈的物种进化史、开展其重要性状遗传基础的研究以及种质资源开发与利用,该团队开展了大口黑鲈基因组研究。结合PacBio单分子实时测序与Hi-C技术,获得了首个高质量染色体水平的大口黑鲈基因组。大口黑鲈基因组大小为964Mb,contig N50和scaffoldN50分别为1.23Mb和36.48Mb。结合PacbioRNA测序一共注释出23,701个基因。
多数鲈形目鱼类单倍型染色体数量为24条,而大口黑鲈却仅有23条。本研究通过比较基因组分析发现其1号染色体为融合染色体的证据:大口黑鲈的1号染色体分别对应于花鲈的20和24号染色体、欧洲鲈的18-21和24-25号染色体,同时在大口黑鲈1号染色体的中上部24Mb处发现了高密度端粒元件区域,提示大口黑鲈1号染色体由2条端部着丝粒染色体融合所成,其中一个着丝粒可能丢失或退化。
论文还探讨了大口黑鲈的咸淡水适应的分子机制。通过收缩与扩张分析、正选择分析和转录组分析,共发现大口黑鲈与离子调控相关基因294个。其中大口黑鲈闭合蛋白基因明显扩张,产生27个家族成员共68个拷贝,该家族成员在调节细胞间紧密连接,离子与水的进出、维持渗透压稳定中发挥重要作用。大口黑鲈基因组精细图谱的完成,为其种质资源保护与改良、进化历史及重要性状的遗传研究提供了重要的数据基础。
该期刊为生物化学与分子生物学、生态学领域国际顶级期刊,中科院JCR分区生物学1区(Q1),IF为6.286。该研究受现代农业产业技术体系专项资金(CARS-46)和水科院科技创新团队项目的支持。中国水产科学研究院珠江水产研究所为论文第一完成单位,孙成飞博士与李佳博士为论文共同第一作者,叶星研究员和石琼研究员为论文共同通讯作者。
文章链接:https://onlinelibrary.wiley.com/doi/epdf/10.1111/1755-0998.13256
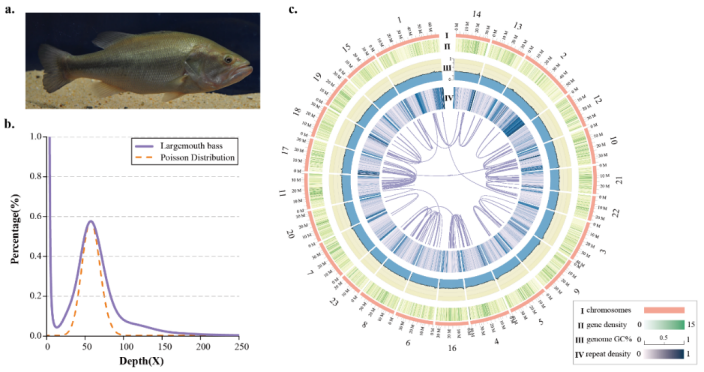
Fig. 1The chromosome-level genome assembly and circos atlas of the largemouth bass.
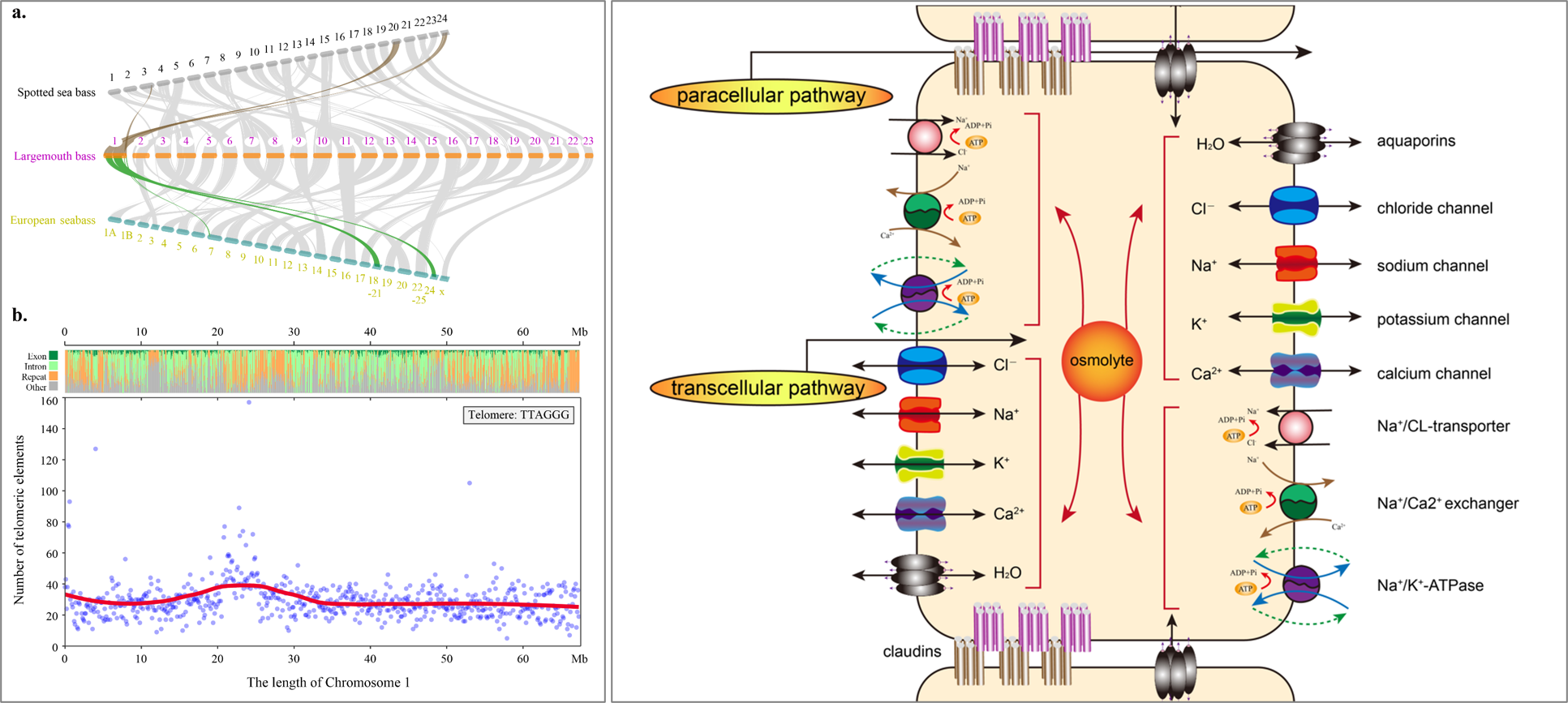
Fig. 2Chromosomal fusion in the Chr1(Left)and the proposed gene network map for ion regulation in LMB(Right)


